Durante a reação de PCR, alguns fatores de interferência são frequentemente encontrados.
Devido à alta sensibilidade da PCR, a contaminação é considerada um dos fatores mais importantes que afetam os resultados da PCR e pode produzir resultados falso-positivos.
Igualmente críticas são as diversas fontes que levam a resultados falso-negativos. Se uma ou mais partes essenciais da mistura de PCR ou a própria reação de amplificação forem inibidas ou interferidas, o ensaio diagnóstico pode ser prejudicado. Isso pode levar à redução da eficiência e até mesmo a resultados falso-negativos.
Além da inibição, a perda da integridade do ácido nucleico alvo pode ocorrer devido às condições de transporte e/ou armazenamento antes da preparação da amostra. Em particular, altas temperaturas ou armazenamento inadequado podem causar danos às células e aos ácidos nucleicos. A fixação de células e tecidos e a inclusão em parafina são causas bem conhecidas de fragmentação do DNA e um problema persistente (ver Figuras 1 e 2). Nesses casos, mesmo o isolamento e a purificação ideais não ajudarão.
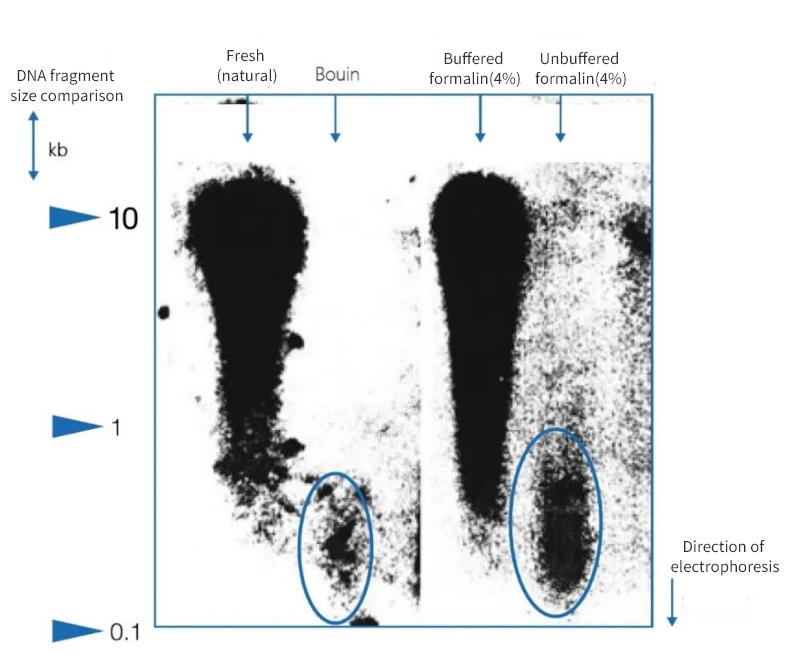
Figura 1 | Efeito da imobilização na integridade do DNA
A eletroforese em gel de agarose mostrou que a qualidade do DNA isolado de cortes de parafina de autópsias variou consideravelmente. DNA com diferentes comprimentos médios de fragmentos estava presente nos extratos, dependendo do método de fixação. O DNA foi preservado apenas quando fixado em amostras nativas congeladas e em formalina neutra tamponada. O uso de um fixador de Bouin fortemente ácido ou formalina não tamponada contendo ácido fórmico resultou em uma perda significativa de DNA. A fração restante é altamente fragmentada.
À esquerda, o comprimento dos fragmentos é expresso em pares de quilobases (kbp)
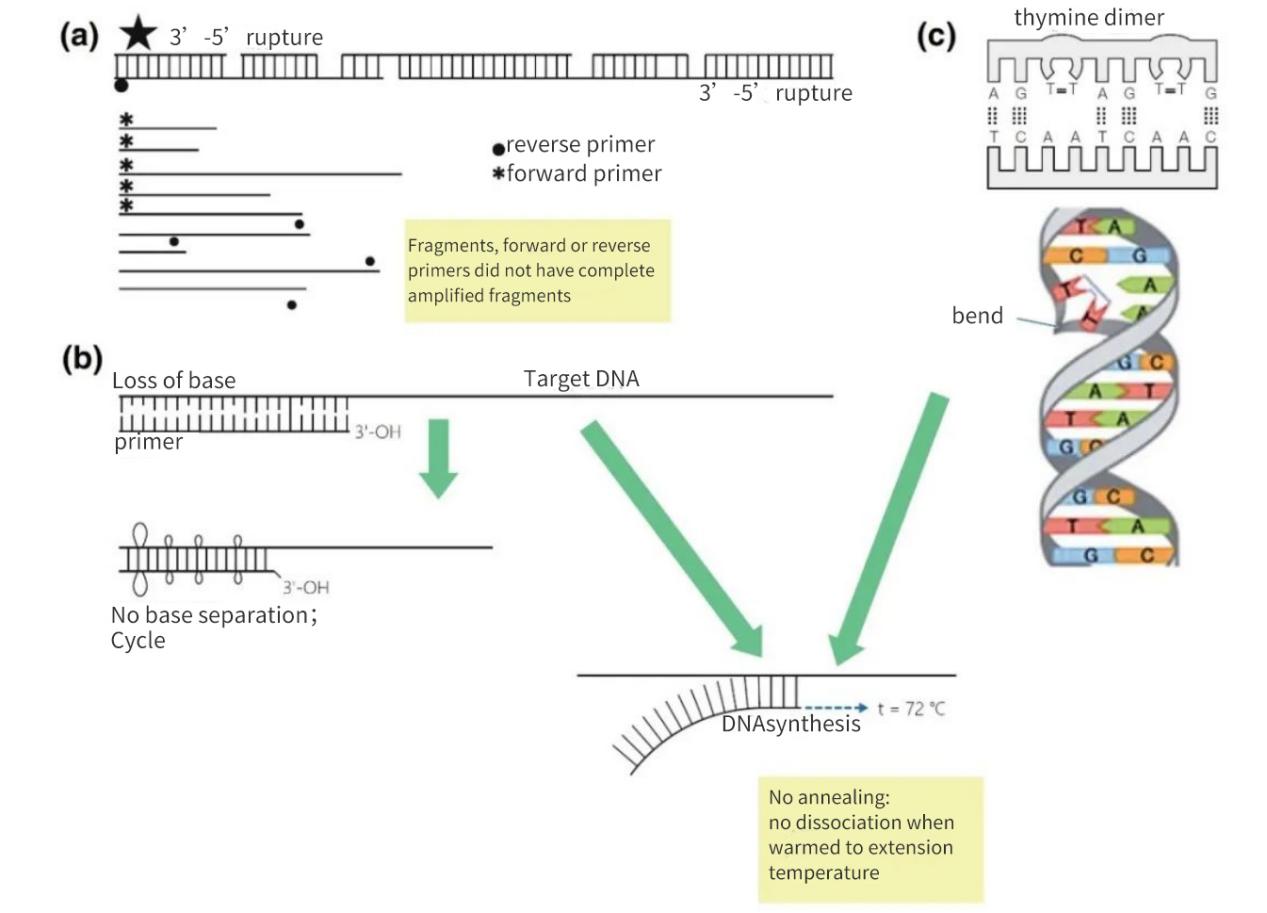
Figura 2 | Perda de integridade de alvos de ácido nucleico
(a) Uma lacuna de 3′-5′ em ambas as fitas resultará em uma quebra no DNA alvo. A síntese de DNA ainda ocorrerá no pequeno fragmento. No entanto, se um sítio de anelamento de primer estiver ausente no fragmento de DNA, ocorrerá apenas amplificação linear. No caso mais favorável, os fragmentos podem ressaturar-se mutuamente, mas os rendimentos serão pequenos e abaixo dos níveis de detecção.
(b) A perda de bases, principalmente devido à depurinação e à formação de dímeros de timidina, leva à diminuição do número de ligações de hidrogênio e à diminuição da Tm. Durante a fase de aquecimento prolongada, os primers se desprenderão do DNA da matriz e não se hibridarão, mesmo em condições menos rigorosas.
(c) Bases de timina adjacentes formam um dímero TT.
Outro problema comum que ocorre frequentemente em diagnósticos moleculares é a liberação abaixo do ideal de ácidos nucleicos alvo em comparação com a extração com fenol-clorofórmio. Em casos extremos, isso pode estar associado a falsos negativos. A lise por fervura ou a digestão enzimática de resíduos celulares podem economizar muito tempo, mas esse método frequentemente resulta em baixa sensibilidade da PCR devido à liberação insuficiente de ácidos nucleicos.
Inibição da atividade da polimerase durante a amplificação
Em geral, a inibição é usada como um conceito de contêiner para descrever todos os fatores que levam a resultados de PCR abaixo do ideal. Em um sentido estritamente bioquímico, a inibição limita-se à atividade da enzima, ou seja, reduz ou impede a conversão substrato-produto por meio da interação com o sítio ativo da DNA polimerase ou seu cofator (por exemplo, Mg2+ para a Taq DNA polimerase).
Componentes na amostra ou vários tampões e extratos contendo reagentes podem inibir diretamente a enzima ou capturar seus cofatores (por exemplo, EDTA), inativando assim a polimerase e, por sua vez, levando a resultados de PCR diminuídos ou falsos negativos.
No entanto, muitas interações entre componentes da reação e ácidos nucleicos contendo o alvo também são designadas como "inibidores de PCR". Uma vez que a integridade da célula é rompida pelo isolamento e o ácido nucleico é liberado, podem ocorrer interações entre a amostra e a solução e a fase sólida circundantes. Por exemplo, "scavengers" podem se ligar a DNA de fita simples ou dupla por meio de interações não covalentes e interferir no isolamento e na purificação, reduzindo o número de alvos que eventualmente chegam ao recipiente de reação de PCR.
Em geral, os inibidores de PCR estão presentes na maioria dos fluidos corporais e reagentes usados em testes de diagnóstico clínico (ureia na urina, hemoglobina e heparina no sangue), suplementos alimentares (componentes orgânicos, glicogênio, gordura, íons Ca2+) e componentes do meio ambiente (fenóis, metais pesados).
| Inibidores | Fonte |
| Íons de cálcio | Leite, tecido ósseo |
| Colágeno | Tecido |
| Sais biliares | Fezes |
| Hemoglobina | Em sangue |
| Hemoglobina | Amostras de sangue |
| Ácido húmico | Solo, planta |
| Sangue | Sangue |
| Lactoferrina | Sangue |
| melanina (europeia) | Pele, cabelo |
| Mioglobina | Tecido muscular |
| Polissacarídeos | Planta, fezes |
| Protease | Leite |
| Ureia | Urina |
| Mucopolissacarídeo | Cartilagem, membranas mucosas |
| Lignina, celulose | Plantas |
Inibidores de PCR mais prevalentes podem ser encontrados em bactérias e células eucarióticas, DNA não alvo, macromoléculas de ligação a DNA de matrizes teciduais e equipamentos de laboratório, como luvas e plásticos. A purificação de ácidos nucleicos durante ou após a extração é o método preferido para a remoção de inibidores de PCR.
Atualmente, diversos equipamentos de extração automatizados podem substituir muitos protocolos manuais, mas a recuperação e/ou purificação de 100% dos alvos nunca foi alcançada. Inibidores potenciais podem ainda estar presentes nos ácidos nucleicos purificados ou podem já ter feito efeito. Existem diferentes estratégias para reduzir o impacto dos inibidores. A escolha da polimerase apropriada pode ter um impacto significativo na atividade do inibidor. Outros métodos comprovados para reduzir a inibição da PCR incluem o aumento da concentração da polimerase ou a aplicação de aditivos como BSA.
A inibição das reações de PCR pode ser demonstrada pelo uso do controle de qualidade do processo interno (IPC).
Deve-se tomar cuidado para remover todos os reagentes e outras soluções do kit de extração, como etanol, EDTA, CETAB, LiCl, GuSCN, SDS, isopropanol e fenol, do isolado de ácido nucleico por meio de uma etapa de lavagem completa. Dependendo da concentração, eles podem ativar ou inibir a PCR.
Horário de publicação: 19 de maio de 2023
 中文网站
中文网站