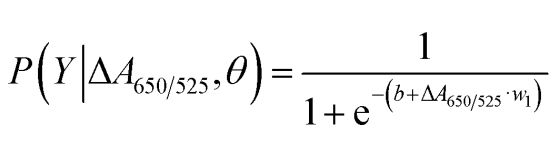A detecção precoce do câncer com base na biópsia líquida é uma nova direção de detecção e diagnóstico do câncer proposta pelo Instituto Nacional do Câncer dos EUA nos últimos anos, com o objetivo de detectar o câncer precoce ou mesmo lesões pré-cancerosas.Tem sido amplamente utilizado como um novo biomarcador para o diagnóstico precoce de várias doenças malignas, incluindo câncer de pulmão, tumores gastrointestinais, gliomas e tumores ginecológicos.
O surgimento de plataformas para identificar biomarcadores de paisagem de metilação (Methylscape) tem o potencial de melhorar significativamente a triagem precoce existente para o câncer, colocando os pacientes no estágio tratável mais precoce.
Recentemente, os pesquisadores desenvolveram uma plataforma de detecção simples e direta para detecção de cenário de metilação com base em nanopartículas de ouro decoradas com cisteamina (Cyst/AuNPs) combinadas com um biossensor baseado em smartphone que permite a triagem precoce rápida de uma ampla gama de tumores.A triagem precoce para leucemia pode ser realizada dentro de 15 minutos após a extração do DNA de uma amostra de sangue, com uma precisão de 90,0%.O título do artigo é Detecção rápida de DNA de câncer no sangue humano usando AuNPs com cisteamina e um smartphone habilitado para aprendizado de máquina。
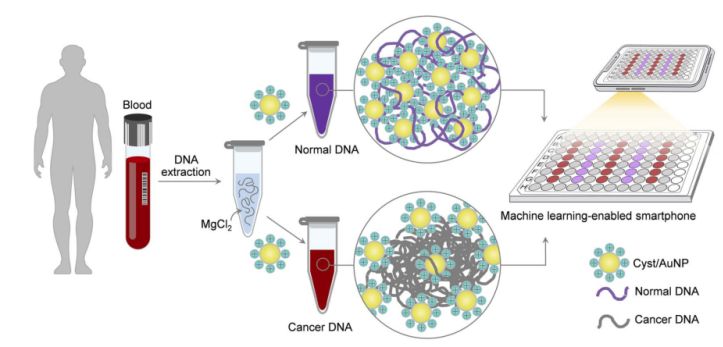
Figura 1. Uma plataforma de detecção simples e rápida para triagem de câncer via componentes Cyst/AuNPs pode ser realizada em duas etapas simples.
Isso é mostrado na Figura 1. Primeiro, uma solução aquosa foi usada para dissolver os fragmentos de DNA.Cyst/AuNPs foram então adicionados à solução mista.DNA normal e maligno têm diferentes propriedades de metilação, resultando em fragmentos de DNA com diferentes padrões de automontagem.O DNA normal agrega-se vagamente e eventualmente agrega Cyst/AuNPs, o que resulta na natureza deslocada para o vermelho de Cyst/AuNPs, de modo que uma mudança na cor de vermelho para roxo pode ser observada a olho nu.Em contraste, o perfil de metilação único do DNA do câncer leva à produção de grupos maiores de fragmentos de DNA.
Imagens de placas de 96 poços foram tiradas usando uma câmera de smartphone.O DNA do câncer foi medido por um smartphone equipado com aprendizado de máquina em comparação com métodos baseados em espectroscopia.
Triagem de câncer em amostras de sangue reais
Para estender a utilidade da plataforma de detecção, os investigadores aplicaram um sensor que distinguiu com sucesso entre DNA normal e cancerígeno em amostras de sangue reais.padrões de metilação em sítios CpG regulam epigeneticamente a expressão gênica.Em quase todos os tipos de câncer, foram observadas alterações na metilação do DNA e, portanto, na expressão de genes que promovem a tumorigênese.
Como modelo para outros tipos de câncer associados à metilação do DNA, os pesquisadores usaram amostras de sangue de pacientes com leucemia e controles saudáveis para investigar a eficácia do cenário de metilação na diferenciação de cânceres leucêmicos.Este biomarcador de cenário de metilação não apenas supera os métodos de triagem rápida de leucemia existentes, mas também demonstra a viabilidade de estender a detecção precoce de uma ampla gama de cânceres usando este ensaio simples e direto.
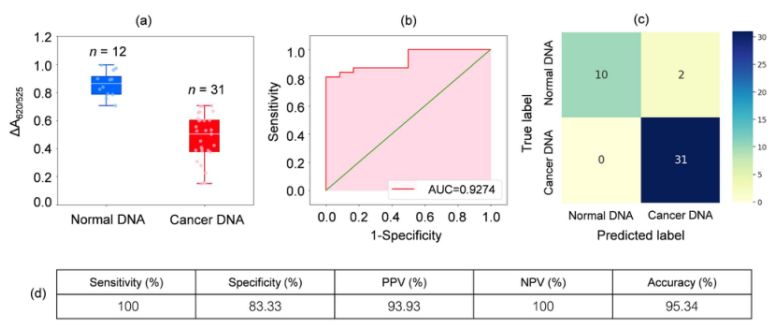
O DNA de amostras de sangue de 31 pacientes com leucemia e 12 indivíduos saudáveis foi analisado.conforme mostrado no box plot da Figura 2a, a absorbância relativa das amostras de câncer (ΔA650/525) foi menor do que a do DNA de amostras normais.isso se deveu principalmente à hidrofobicidade aumentada, levando à agregação densa do DNA do câncer, o que impediu a agregação de cisto/AuNPs.Como resultado, essas nanopartículas foram completamente dispersas nas camadas externas dos agregados de câncer, o que resultou em uma dispersão diferente de Cyst/AuNPs adsorvidos em agregados de DNA normais e de câncer.As curvas ROC foram então geradas variando o limite de um valor mínimo de ΔA650/525 a um valor máximo.
Figura 2.(a) Valores relativos de absorbância de soluções de cisto/AuNPs mostrando a presença de DNA normal (azul) e de câncer (vermelho) sob condições otimizadas
(DA650/525) de box plots;(b) Análise ROC e avaliação de testes diagnósticos.(c) Matriz de confusão para o diagnóstico de pacientes normais e com câncer.(d) Sensibilidade, especificidade, valor preditivo positivo (VPP), valor preditivo negativo (VPN) e acurácia do método desenvolvido.
Conforme mostrado na Figura 2b, a área sob a curva ROC (AUC = 0,9274) obtida para o sensor desenvolvido apresentou alta sensibilidade e especificidade.Como pode ser visto no box plot, o ponto mais baixo que representa o grupo de DNA normal não está bem separado do ponto mais alto que representa o grupo de DNA do câncer;portanto, a regressão logística foi usada para diferenciar entre os grupos normais e com câncer.Dado um conjunto de variáveis independentes, estima a probabilidade de ocorrência de um evento, como um câncer ou um grupo normal.A variável dependente varia entre 0 e 1. O resultado é, portanto, uma probabilidade.Determinamos a probabilidade de identificação do câncer (P) com base em ΔA650/525 como segue.
onde b=5,3533,w1=-6,965.Para a classificação da amostra, uma probabilidade inferior a 0,5 indica uma amostra normal, enquanto uma probabilidade de 0,5 ou superior indica uma amostra de câncer.A Figura 2c mostra a matriz de confusão gerada a partir da validação cruzada leave-it-alone, que foi usada para validar a estabilidade do método de classificação.A Figura 2d resume a avaliação do teste diagnóstico do método, incluindo sensibilidade, especificidade, valor preditivo positivo (VPP) e valor preditivo negativo (VPN).
Biossensores baseados em smartphones
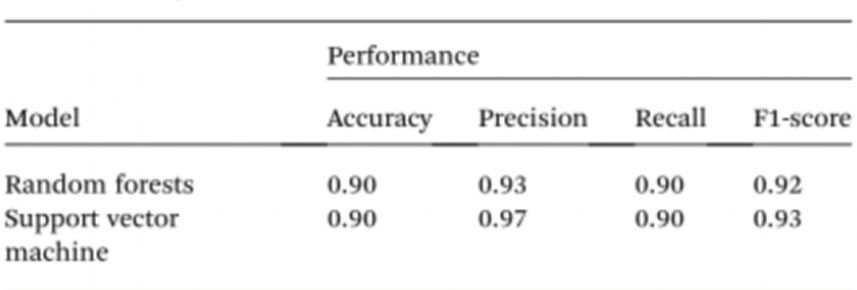
Para simplificar ainda mais o teste de amostras sem o uso de espectrofotômetros, os pesquisadores usaram inteligência artificial (IA) para interpretar a cor da solução e distinguir entre indivíduos normais e cancerosos.Diante disso, a visão computacional foi usada para traduzir a cor da solução Cyst/AuNPs em DNA normal (roxo) ou DNA canceroso (vermelho) usando imagens de placas de 96 poços tiradas por meio de uma câmera de celular.A inteligência artificial pode reduzir custos e melhorar a acessibilidade na interpretação da cor das soluções de nanopartículas, e sem o uso de qualquer acessório de smartphone de hardware óptico.Finalmente, dois modelos de aprendizado de máquina, incluindo Random Forest (RF) e Support Vector Machine (SVM), foram treinados para construir os modelos.ambos os modelos RF e SVM classificaram corretamente as amostras como positivas e negativas com uma precisão de 90,0%.Isso sugere que o uso de inteligência artificial em biossensores baseados em telefones celulares é bem possível.
Figura 3.(a) Classe alvo da solução registrada durante o preparo da amostra para a etapa de aquisição da imagem.(b) Exemplo de imagem tirada durante a etapa de aquisição da imagem.(c) Intensidade da cor da solução de cisto/AuNPs em cada poço da placa de 96 poços extraída da imagem (b).
Usando Cyst/AuNPs, os pesquisadores desenvolveram com sucesso uma plataforma de detecção simples para detecção de paisagem de metilação e um sensor capaz de distinguir o DNA normal do DNA do câncer ao usar amostras de sangue reais para triagem de leucemia.O sensor desenvolvido demonstrou que o DNA extraído de amostras de sangue reais foi capaz de detectar rapidamente e com baixo custo pequenas quantidades de DNA de câncer (3nM) em pacientes com leucemia em 15 minutos e mostrou uma precisão de 95,3%.Para simplificar ainda mais o teste de amostras, eliminando a necessidade de um espectrofotômetro, o aprendizado de máquina foi usado para interpretar a cor da solução e diferenciar entre indivíduos normais e cancerosos usando uma fotografia de telefone celular, e a precisão também foi alcançada em 90,0%.
Referência: DOI: 10.1039/d2ra05725e
Horário da postagem: 18 de fevereiro de 2023
 中文网站
中文网站